头秃元凶「真面目」首次被揭穿,鹅厂程序员立功了|Nature子刊
还揭示了非那雄胺防脱发机制
贾浩楠 鱼羊 发自 凹非寺
量子位 报道 | 公众号 QbitAI
好消息,好消息。
打工人的噩梦,脱发的元凶,它的高清大头照现在科学家扒!出!来!了!
就是它,脱发之源SRD5A2(II型5a还原酶)↑↑↑
这是SRD5A2的高分辨率三维蛋白质结构首次被破解,并且分辨率达到了2.8埃(1埃=10-7毫米)。
也就是说,疗效更好的防脱新药,现在有了研发的关键靶点。

同时,知名治脱药物「非那雄胺」的防脱机制也在这项研究中被揭示。
这一来自南科大、匹兹堡大学、新加坡A*STAR研究所和腾讯AI Lab的成果已经登上了Nature Communications。
其中,AI还立下了一桩大功。
具体如何,快来一起围观:
「秃如其来」的真凶
熬夜秃,不熬夜的,也秃了。
引起脱发的原因很多。
但对于二三十岁就秃了头的年轻人群来说,攻击范围最广、最顽固的「元凶」,是雄性激素脱发。
引起雄脱的原因,就是毛囊杀手——DHT(二氢睾酮)。
人体内的睾酮(男女体内都有)在SRD5A2的作用下,可进一步代谢为与毛囊雄激素受体结合能力更强的DHT。
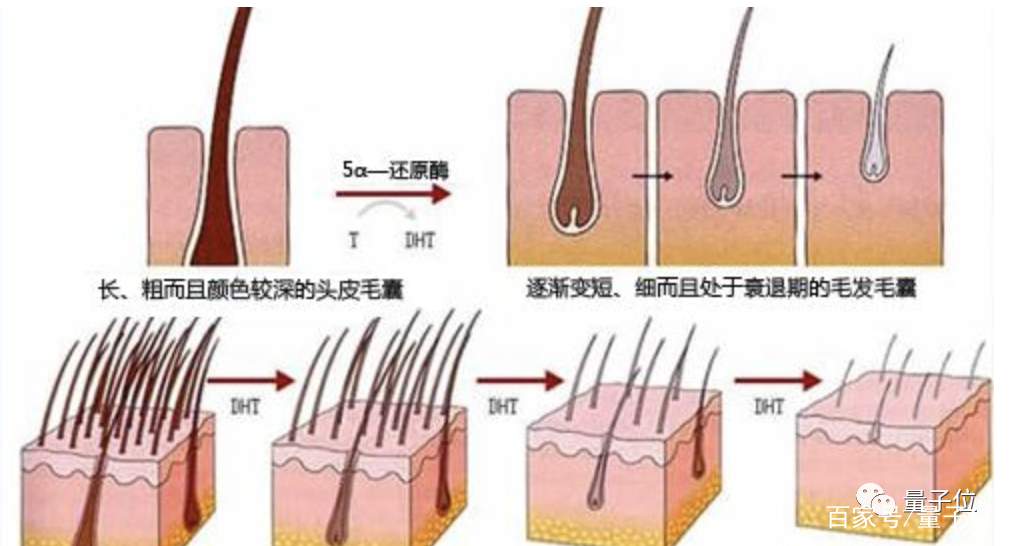
DHT进入细胞后,会对代谢系统产生作用,使作为能量源的ATP无法产生,从而无法进行毛发的蛋白合成。毛母细胞因此失去活力,角质化形成休止期毛发,大概三个月后就会脱落。
在对抗脱发的斗争中,人们早就了解到SRD5A2还原酶是导致雄脱的幕后「真凶」,也偶然间发现了治疗前列腺增生的非那雄胺有抑制SRD5A2的作用,并将它作为治疗雄脱的主要药物。

但直到最近,SRD5A2的高分辨率蛋白质结构才首次被破解:
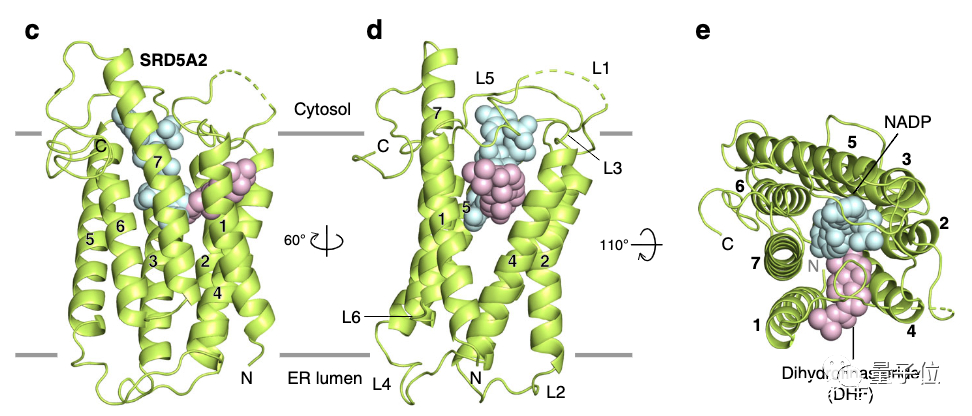
与细菌甾醇还原酶MaSR1不同的是,SRD5A2具有独特的七次跨膜结构,由6个环连接:
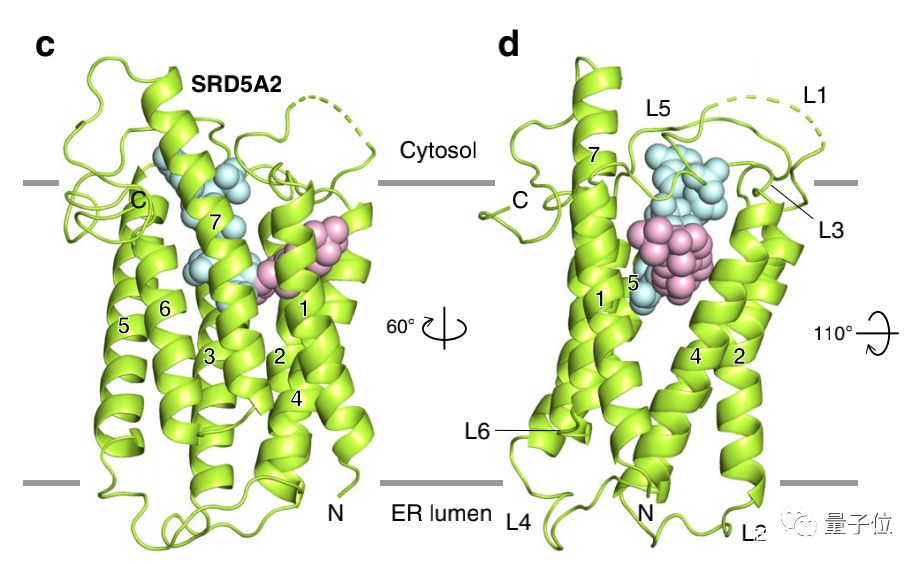
团队根据羟基侧富含的正电荷残基,将羧基末端的环(C-loop)面对细胞质,将氨基末端环(N-loop)面对内质网腔。
此外,氨基末端残基C5N与环4(L4)中的C133L4形成二硫键,这表明氨基端侧面对的正是内置网腔位置,这是因为细胞质部分拥有还原性环境。
至于为什么非那雄胺(Finasteride)能够抑制SRD5A2,首先要从SRD5A2的作用讲起。
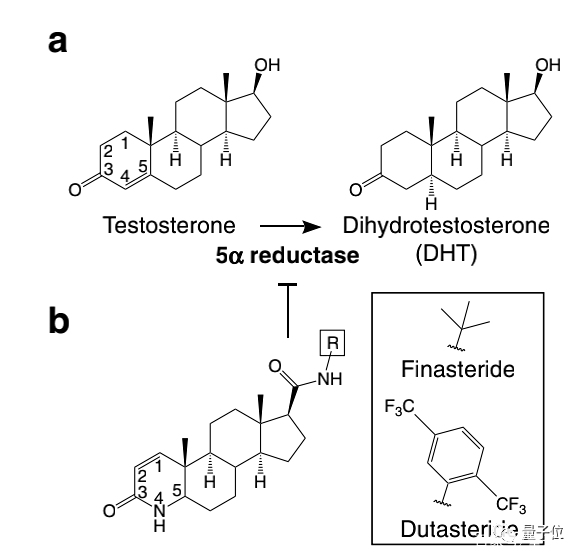
图a表示的是SRD5A2通过还原睾酮的双键,将睾酮转化为二氢睾酮。
而非那雄胺的作用,则是通过本身结构中的侧链(图b中的R基),连接到SRD5A2的酰胺基链,形成二氢非那雄胺(DHF),提前「预支」掉SRD5A2的还原作用。
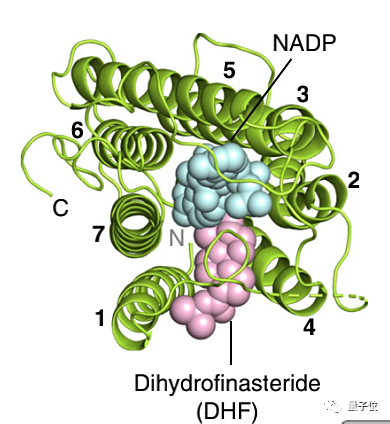
在蛋白质结构3D示意图中,粉红色标出的部分就是二氢非那雄胺,而与之链接的青色部分,是SRD5A2的酰胺基链,也就是通常还原性辅酶II中的主要功能性结构NADP。
你可能会问,既然已经发现了针对雄性激素脱发的有效治疗药物,再做如此深入的基础研究,有什么重要性?
其实,了解蛋白质的微观结构,能够更好地摸清它的性质,当然就能根据这些性质针对性地利用。
比如,目前的非那雄胺并不是治疗雄性激素脱发的完美方案。
首先,非那雄胺具有一定的副作用风险,比如有概率引起男性阳痿,按照美国FDA记录的数据,这个概率在2%左右。
而且,停药之后,没了「预支」SRD5A2还原作用的非那雄胺,脱发很可能复发。
除了引起阳痿、疗效反弹,非那雄胺还有一个很大的局限性,即女性不能使用,一是因为确实没什么效果,二是会对生理周期产生影响。
而了解了SRD5A2的结构和非那雄胺作用机制,可以让科研人员模仿类似机制研制出副作用更轻的替代药品,或者从基因侧入手,抑制SRD5A2的表达。
不仅如此,对于SRD5A2活性失调引发的各种疾病,这一成果也有着重要的参考价值。
如何破解SRD5A2「真面目」
所以,研究人员们具体是如何得到SRD5A2的高分辨率结构信息的呢?
「从头折叠」
想要破解SRD5A2的真面目,主要的技术难点在于,它具有独特的7次跨膜结构,与人类全部已知结构的蛋白质差异较大,难以通过常用的「模板建模」方法获得初始构型,来解析晶体数据。
于是,研究人员基于腾讯 AI Lab开发的tFold工具,采用了「从头折叠(de novo folding)」的方法。该方法可以不依赖于模板来预测蛋白质结构。
具体而言,参照蛋白质结构预测算法trRosetta,首先,根据序列比对文件来预测氨基酸之间的距离和朝向分布,并将其转化为势能。
然后,用这一结果作为约束条件,与粗粒度的能量优化一起输入蛋白质建模工具Rosetta。
最后,根据Rosetta能量,选择满足约束条件的最佳结构。
在这个实现过程中,tFold工具提供了三项技术辅助,进一步提高了从头折叠方法蛋白结构预测的精度:
腾讯 AI Lab研发的多数据来源融合(multi-source fusion)技术,用来挖掘多组多序列联配(multiplesequence alignment, MSA)中的共进化信息。
主要是借助多个不同的MSA(多重序列对比)搜索工具,在不同的参数、不同来源的蛋白序列数据库下,得到的具有多样性的MSA,然后再采用一种深度学习技术将其包含的共进化信息给融合起来。
深度交叉注意力残差网络(deep cross-attention residual network,DCARN),能大幅提高残基对距离、取向矩阵等重要蛋白2D结构信息的预测精度。
其核心在于利用互相交叉的2D注意力机制,来更有效的捕捉远程的残基对相互作用。这种2D注意力机制的表现能力,比通常的深度卷积神经网络更强。
模板辅助自由建模(Template-basedFree Modeling, TBFM)方法,将自由建模(Free Modeling, FM)和模板建模(Template-based Modeling, TBM)生成的3D模型中的结构信息加以有效融合,从而提高最终3D建模的准确性。
其核心是,将TBM得到的3D模型中的残基对2D结构信息,以一种输入特征的方式,添加到预测2D结构特征的深度交叉注意力残差网络当中去,更好的帮助该网络进行2D结构特征的预测。
这样,即便在某些情况下TBM得到的3D模型精度不高,但其中的部分残基对结构信息(例如某些很保守的距离,或者取向)依然可以被有效的利用,从而更好的帮助FM进行精确的折叠。
值得一提的是,预测SRD5A2这个蛋白,tFold只花费了2小时左右的时间。
X射线数据收集和结构测定
研究人员还在美国阿贡国家实验室收集到了SRD5A2蛋白质晶体的X射线衍射数据。并使用HKL2000软件对来自5个晶体的数据集进行了处理和合并。
为了确定晶体结构的相位,研究人员采用从头折叠结构模型作为分子置换的搜索模型,使得SRD5A2的结构精度达到了2.8埃。
One More Thing
现在,tFold公测版本已通过腾讯「云深智药」平台官网对外开放,蛋白质结构预测、虚拟筛选、分子生成、ADMET预测等功能都可以免费使用。
量子位已经体验了一把,效果大致是酱婶的,预测速度也挺快:

如果你也感兴趣,不妨亲自一试。
最后,祝各位打工人头发健康~
传送门:
论文地址:
https://www.nature.com/articles/s41467-020-19249-z#Sec10
腾讯云深智药官网:
https://drug.ai.tencent.com/cn
参考链接:
https://mp.weixin.qq.com/s/DQdfJWZKtnGcoYcEpDZn_A
https://yanglab.nankai.edu.cn/trRosetta/
— 完 —
- 马斯克冲刺机器人量产,果断停产特斯拉豪华车型!2026年资本支出将“非常大”2026-01-29
- 那个用半成品刷爆SOTA的Qwen3超大杯推理版,现在正式上线2026-01-26
- “DeepSeek-V3基于我们的架构打造”,欧版OpenAI CEO逆天发言被喷了2026-01-26
- 斯坦福「返老还童」新研究:无需干细胞,逆转关节损伤和老化2026-01-25