用AIGC搞药物发现,大分子那种,来自Bengio核心团队教授唐建新成果
改变AlphaFold传统
衡宇 发自 凹非寺
量子位 | 公众号 QbitAI
又一名AI大牛,为AI制药领域带来了新进展——
AlphaFold2仅仅是蛋白质结构建模迈出的第一步,现在可以基于结构进行蛋白质设计了!
最近在智源大会AI+生命科学论坛上,来自Mila实验室的教授唐建介绍了团队的最新成果,一同受邀参会的还有诺贝尔化学奖得主Arieh Warshel等知名学者。
相关论文《Protein Representation Learning by Geometric Structure Pretraining》已经被顶会ICLR’23接收,还在GitHub上开源。

研究团队由深度学习三巨头之一Yoshua Bengio的Mila实验室、蒙特利尔大学、剑桥大学、IBM Research等机构组成。
在MILA这个学术界最大AI研究中心之一里,唐建任终身教授,同时也是该实验室创办以来唯一的华人教授。
其研究方向主要包括几何深度学习、深度生成模型、知识图谱以及这些方法在药物发现中的应用。
AI 2.0浪潮下,唐建想做的事,是加速AI参与药物设计逐渐走向从drug discovery(发现自然界存在的drug)到drug design(直接通过AI设计drug分子)的演变。
于是,延续之前的发展路线,所创AI驱动型生物制药公司百奥几何,近期将重点落到构建寻找新分子结构的生成式模型上。
蛋白质大分子的基础模型,搞它!
唐建团队的目标,是利用生成式AI,向药物设计更进一步。
大分子药物(蛋白质、抗体)在生物医药领域扮演着越来越重要的作用,有着广泛的应用前景。
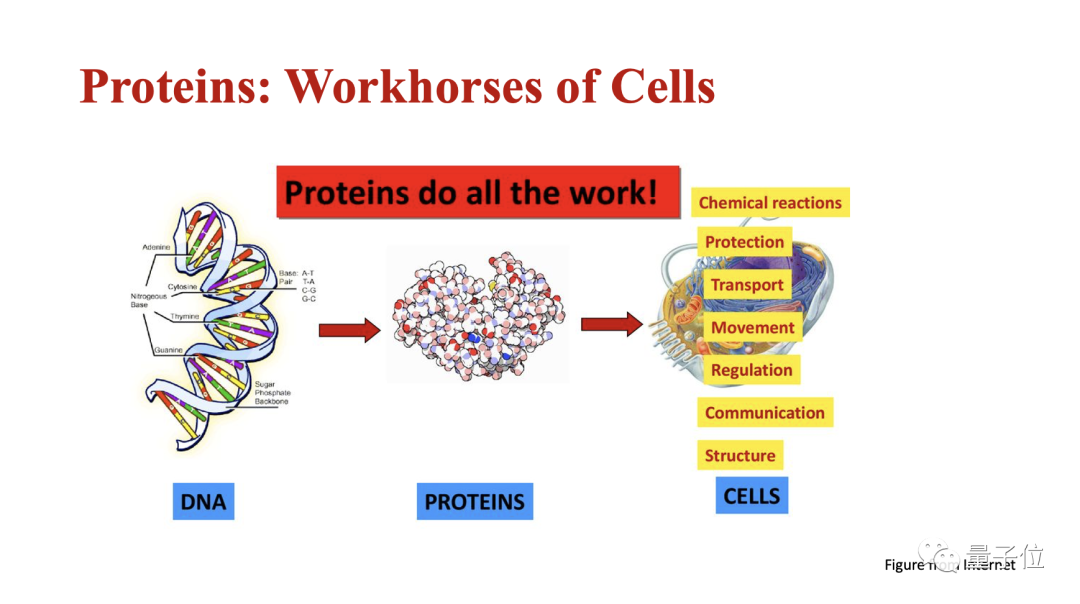
蛋白质的功能有三种不同的表示:一级序列,二级结构,三级空间结构。
大多数已有的蛋白质建模的方法是基于序列的方法。蛋白质的序列决定其3D结构,而结构决定功能。
因此,想要更好地理解蛋白质的功能,从基于结构的角度出发,是一条更好的技术路径。
But!AlphaFold2只是在基于蛋白质结构的建模方向上走出了第一步。
唐建团队做的事,是基于AIGC,进一步推进基于结构的大分子药物设计。

首先基于蛋白质3D结构的表征学习,得到预训练模型,在此基础上就能做更精准的功能预测。
也就是前面介绍到的发表在ICLR’23的论文。
团队首先提出GearNet和GearNet-edge,它们依赖稀疏边缘消息(sparse edge message)的传递,来增强蛋白质图中的序列和结构信息的捕获。
在功能预测和折叠分类任务上,这个方法和最先进的基于序列预测蛋白质表征的方法相当(或优于),而且使用的预训练数据更少。
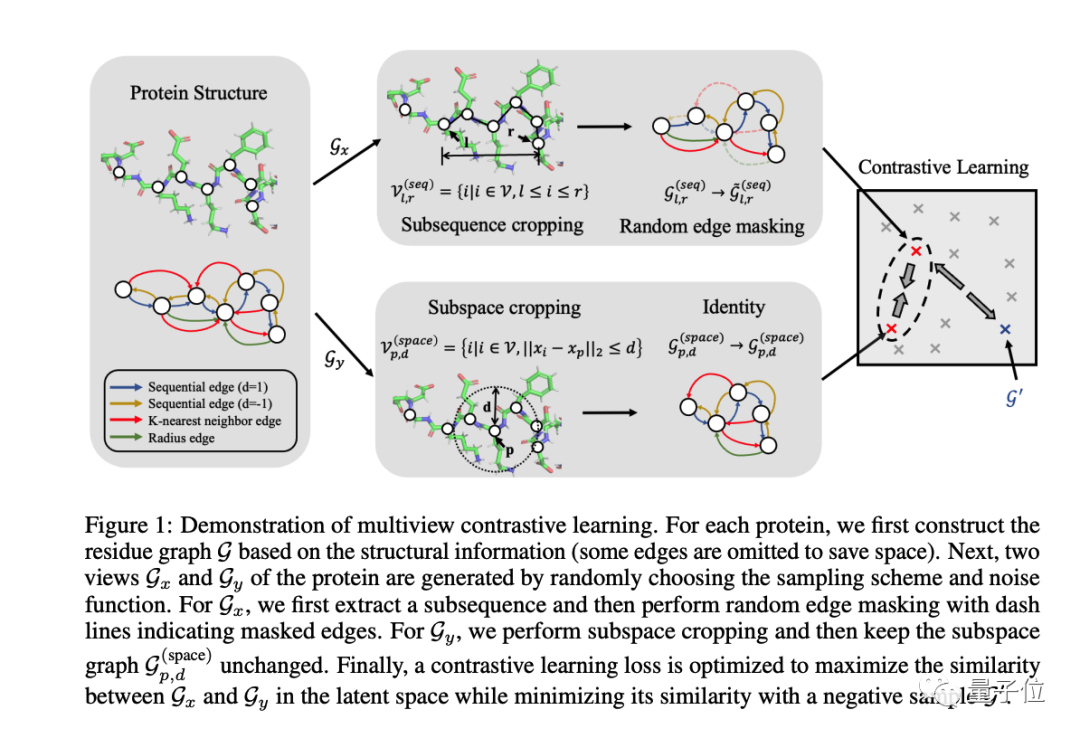
第二步是更精准的结构预测。AlphaFold2主要是对主链进行了预测,对侧链的预测不是很准确。
团队提出扭转扩散模型DiffPack,通过在扭转空间上扩散和去噪,来学习侧链扭转角的联合分布。

侧链是蛋白质结构的构成部分之一,它的差异会带来蛋白质结构和功能的差异。
搞清楚侧链结构,能够更精准地帮助药物设计流程更快更准地抓住药物和受体该在什么点位结合。
DiffPack包含了一个自回归扩散过程,对准确度和速度的限制有所缓解。
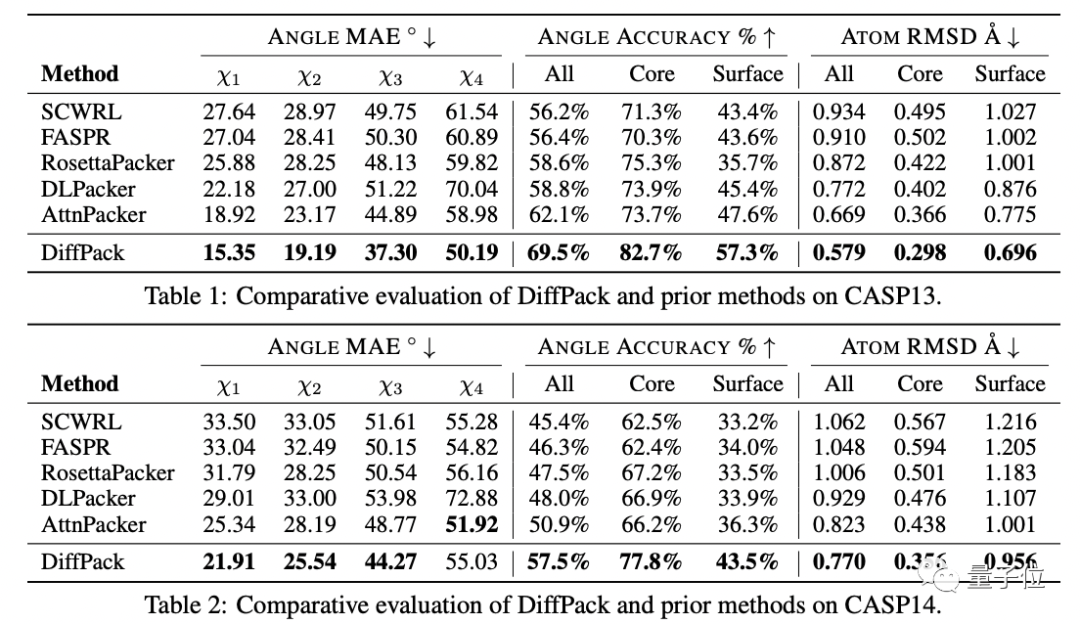
实验结果显示,新方法在CASP13和CASP14上的角度和精度,分别提高了11.9%和13.5%,双双取得SOTA,且模型参数却能减少60倍。
接下来,基于精准结构预测结果,就能更好完成蛋白质设计。
这部分用到了扩散模型。已有的工作如华盛顿知名教授David Baker组的工作RFDiffusion采用的是两阶段算法,首先生成结构,然后根据结构设计蛋白质序列。
唐建团队提出了一个新算法,能够同时对结构和序列进行设计。
也就是说,只需要利用同一个算法,就可以对大分子蛋白质的氨基酸序列和三维结构进行建模和设计,并展示二者之间的相互关系,从而生成具有特定功能的蛋白质。
迄今为止,团队已经实现了抗体CDR Loop结构和序列的生成,还设计了具有指定个数的α螺旋跨膜蛋白。

其实唐建团队这段征程里的主角,即蛋白质3D结构大模型,无论是模型参数和训练数据量都远不如最热门的ChatGPT。
但因其用于垂直领域,还是生物制药领域——人类的语言词汇量多大几千万,而蛋白质的词汇表只有20个——不那么庞大的参数和训练量已经能够满足实际需求,达到比较好的生成效果。
而对训练数据细究来处,有三个源头:生物制药领域公开数据、AlphaFold2等预测出的大分子结构、实验室的自有数据。
来自最后两个实验室的训练数据,被唐建团队视为壁垒之一。
Bengio所领导的实验室核心团队唯一华人教授
2014年,唐建博士毕业于北京大学信息科学技术学院,其研究方向主要包括几何深度学习、深度生成模型等。
而后,唐建远赴美国,在CMU以及密歇根大学进行博士后研究。
2013年,恰逢深度学习兴起,一直专注传统机器学习领域的他在关注到ImageNet、Word2Vec等在深度学习领域取得了非常不错的效果。
“当时我觉得自己的研究遇到了一定的瓶颈,所以也开始想转向深度学习领域。”
唐建称自己做了很多层面的思考,即便是要真的做出研究方向的调整,也想做自己能drive、相对独立的研究,而不是随大流进行“follow”。
最后,他寻找的的机会是用深度学习解决图结构数据,成为国内利用DL研究图结构数据的最早的一批人。
他作为一作的理论工作《Understanding the Limiting Factors of Topic Modeling via Posterior Contraction》获得ML顶级会议ICML 2014的最佳论文。
此后,他在图表示学习领域的工作LINE(《Line: Large-scale information network embedding》)引用次数超5300次,并多次在计算机顶会ICML、NeurlPS担任领域主席。
再后来,因为对“21世纪是生物的世纪”这句话的深信不疑,他的研究逐渐集中在AI和生物交叉领域,在分子性质预测、结构性质预测上进行探索。
2017年12月起,唐建成为Mila实验室(加拿大魁北克省人工智能研究中心)20余名教授中的一员,与深度学习三巨头之一Yoshua Bengio一起,致力于推进AI for Science。
投身创业前,唐建团队已经发布了药物研发机器学习平台TorchDrug。
并在一年后,即2022年9月,联合英伟达、英特尔、IBM等公司,发布了针对大分子药物研发的开源机器学习平台TorchProtein。
平台开源了深度学习对大分子建模的一个通用框架、基于蛋白质三维几何结构的第一个预训练大模型、以及专门用于评价深度学习对蛋白质建模效果的标准数据集。
唐建坦然表示,在接下来的进展中,这些公司也将成为百奥几何在大模型研究中的国际合作伙伴,“这是我们与同行相比的优势所在”。
TorchProtein发布的同时,公司宣布完成千万美元天使轮融资。
现在,唐建白天醉心百奥几何的技术研究,夜晚为远在大洋彼岸的学生们指导。但这样的工作模式并没有让他疲于应对或感到苦恼:
在两者间取得平衡并不是难事,这反而让公司团队更具国际化优势。
- 阶跃星辰不再低调:巨额融资,印奇加入,“1+3”核心决策层浮出水面2026-01-27
- 14万!全球首款家务机器人开卖,OpenAI投资,萌脸翘臀会自己充电2025-10-29
- 将登央视春晚,今年冲击IPO!江苏具身新贵魔法原子联创披露一堆新信息2026-01-24
- 把医疗AI禁锢在严肃区间:百川M3 Plus首创“证据锚定”,幻觉率2.6%刷新全球纪录2026-01-23










