电脑插个“U盘”就能给基因测序,实时查看结果,售价1000美元
把DNA拉丝进纳米小孔测序
博雯 发自 凹非寺
量子位 报道 | 公众号 QbitAI
现在,你也可以像黑客帝国里的Neo一样,查看构成自己和别人的“源代码”了。

方法也非常简单,将体液滴到这样一个大号的“U盘”上:

然后连线插进电脑,再配合软件使用,就能近乎实时地查看你的DNA测序结果了:

毕竟现实中的我们并不生活在矩阵(Matrix)里,也不是由代码组成。
但作为人体生长发育“配方”的DNA序列,某种程度上就相当于一个人的“源代码”了。
而理论上,上述的这一方法甚至能在6小时内对超过1.6亿个碱基进行测序,速度非常快。
这是怎么做到的?
从把DNA“拉丝”并推进一个纳米尺寸大小的孔洞里开始:
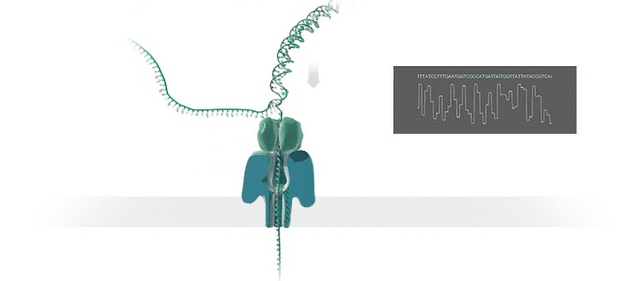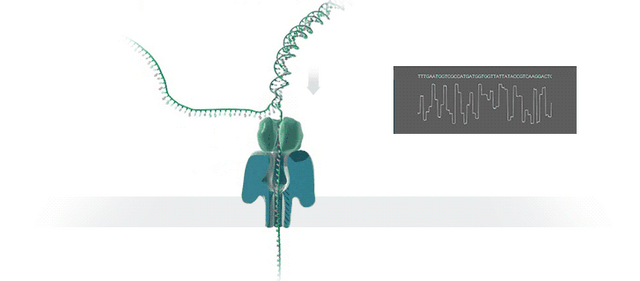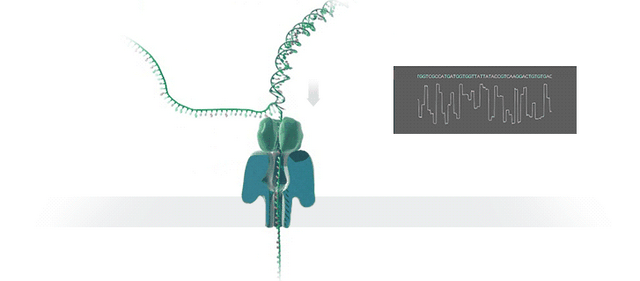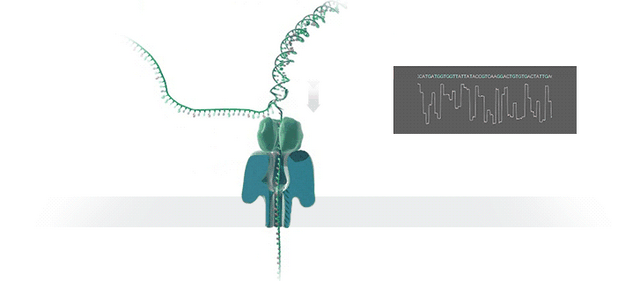
将DNA“拉丝”进洞
这是一种叫做纳米孔测序 (Nanopore sequencing)的技术。
在这种方法里,未知序列的样品会被输送穿过一个直径1纳米的小孔。
以人类体液为样本,需要先利用酶“拉开”DNA分子的“拉链”,让一根单链进入纳米孔:
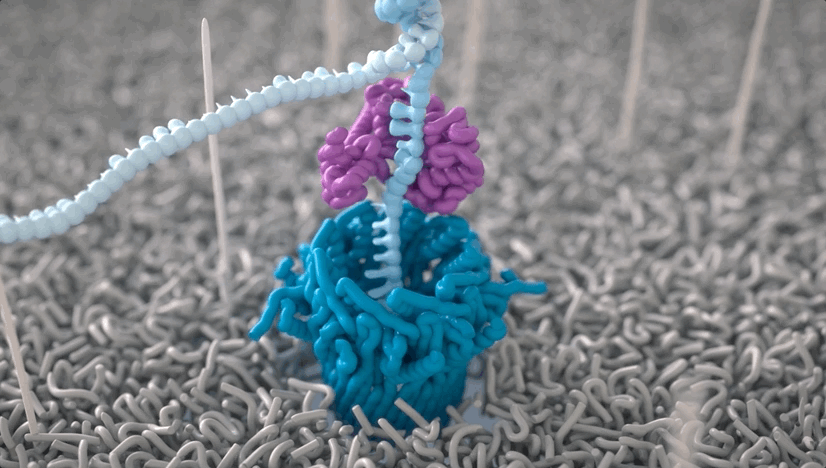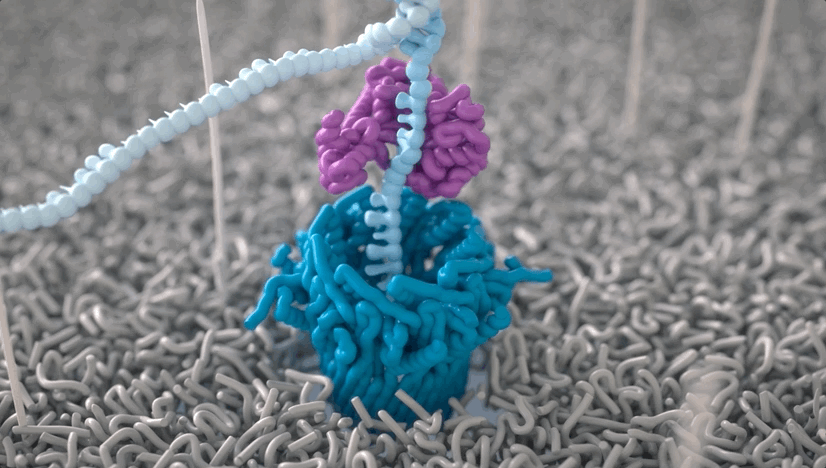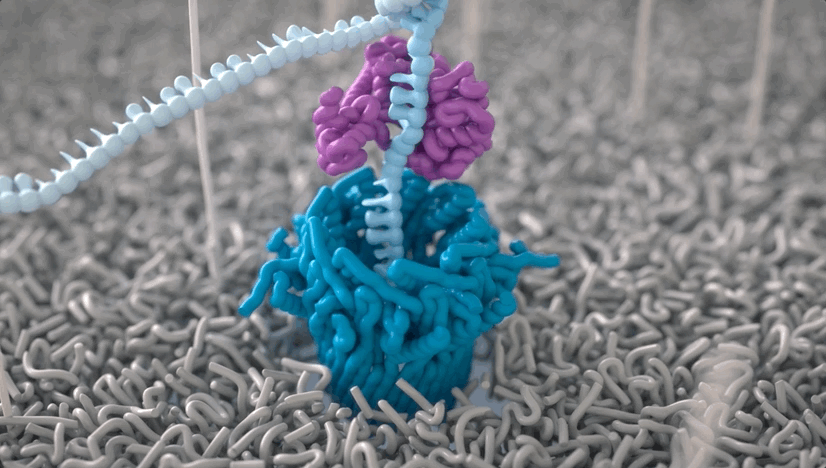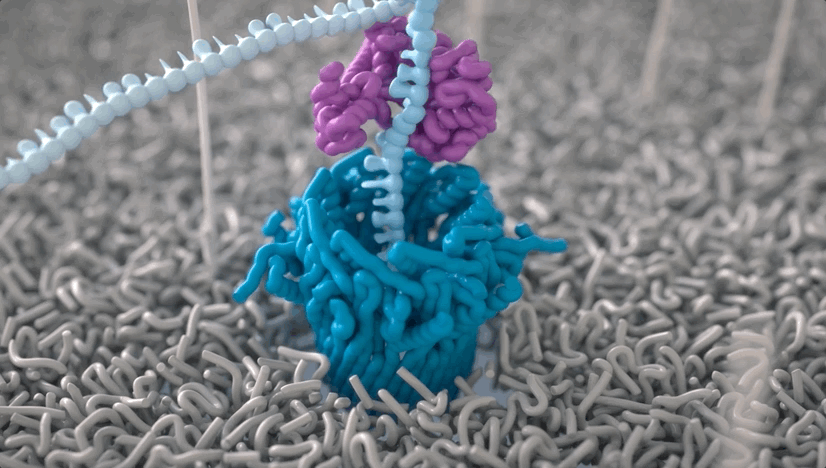
纳米孔所在的生物系统会泡在电解质溶液中,并被施以空间匀强电场。
DNA在核酸外切酶的作用下被迅速逐一切割成单个碱基分子,在电场的驱使下通过纳米孔,此时,就会形成可检测的离子电流。
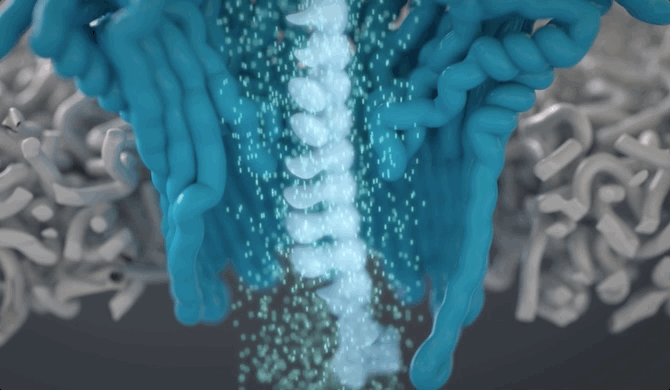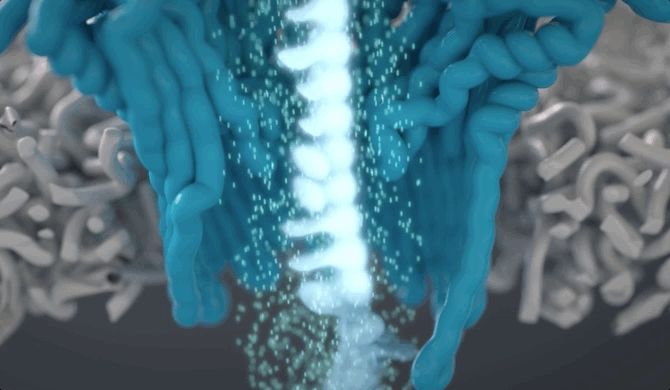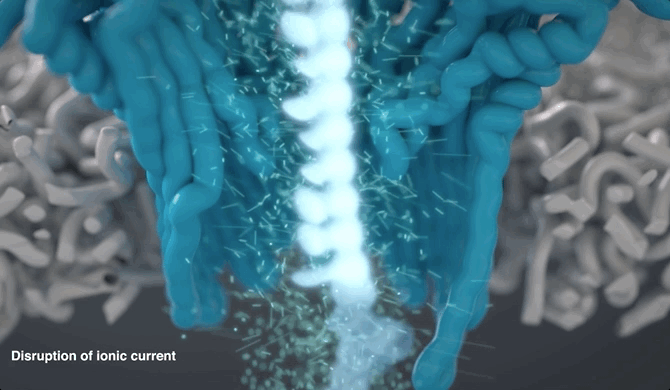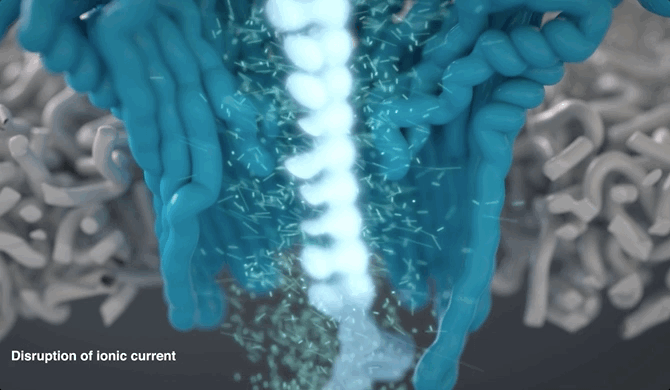
不同碱基的化学性质不同,这也就使得整条DNA链会在穿越纳米孔引起幅度不同的电流变化。
而这种电流信号的强弱,就能反映DNA样本的核酸序列信息。

具体而言,每一个DNA片段都就会返回一个基于核苷酸变化的电压读数。
当然,这种反映并不直观,还需要通过一种类似NLP的算法逻辑,将含有噪声的电信号转化为碱基识别的序列数据。
这种方法比起传统的基因测序方法,比如NGS来说,速度要更快,而且对变异结构的检测也更准确。
给电脑插个“U盘”就能看
而从牛津大学衍生而出的Oxford Nanopore公司,就将上述核心技术打包,塞进了这样一个尺寸10厘米的“U盘”之中:

这一产品叫做MinION,使用时需要将自己的体液滴到指定位置。
然后再用一根线将其与电脑连接,再安装产品相应的软件,此外载无需任何设置。
然后,就能在电脑屏幕上看到数据采集、实时分析和运行反馈、本地基本通信和数据流:
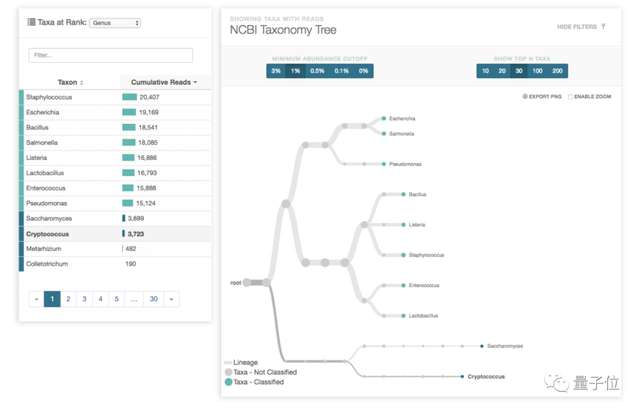
根据官方的数据,理论上最大输出速度可达到420个碱基/秒,而从一个单元格中的样本最多可得到50GB的数据,准确率达到88%以上。
除了对专业的研究人员售卖以外,官方的简介中也表明,“没有大量实验室经验的人群也可使用”。
完整的产品包售价1000美元,折合人民币约6370元。
对于这一产品,有人觉得非常有趣,对于学校或者一些个人开发者来说无疑降低了技术门槛。

也有网友吐槽这只是“听起来很酷”,毕竟除非测序的对象是一个鸡蛋,否则得到的任何序列都只是你全体DNA的一个小子集而已。
最近,还有一位来自霍普金斯大学的教授基于这一产品做了进一步的开发,提出了一个叫做UNCALLED的程序。
在面对大型序列时,这一软件将序列内容与已知的基因序列进行匹配,快速挑选出所需的目标序列。
举个例子,研究人员想要确定一个人是否携带了遗传性癌症相关基因的变异(比如 BRCA1),就可以使用这一软件快速判断当前样本是否值得研究。
论文:
https://www.nature.com/articles/s41587-020-0731-9
开源下载:
https://github.com/skovaka/UNCALLED
参考链接:
[1]https://stackoverflow.blog/2021/12/24/sequencing-your-dna-with-a-usb-dongle-and-open-source-code/
[2]https://news.ycombinator.com/item?id=29695013
[3]https://www.extremetech.com/extreme/190409-minion-usb-stick-gene-sequencer-finally-comes-to-market
- 有道智能学习灯发布,通过“桌面学习分析引擎”实现全球最快指尖查词2022-04-08
- 科学证明:狗勾真的懂你有多累,听到声音0.25秒后就知道你是谁,对人比对狗更亲近2022-04-14
- 在M1芯片上跑原生Linux:编译速度比macOS还快40%2022-04-05
- 小学生们在B站讲算法,网友:我只会阿巴阿巴2022-03-28