谷歌DeepMind再登Science:用AI预测基因突变致病性,PK人类专家=89:0.1 | 开源
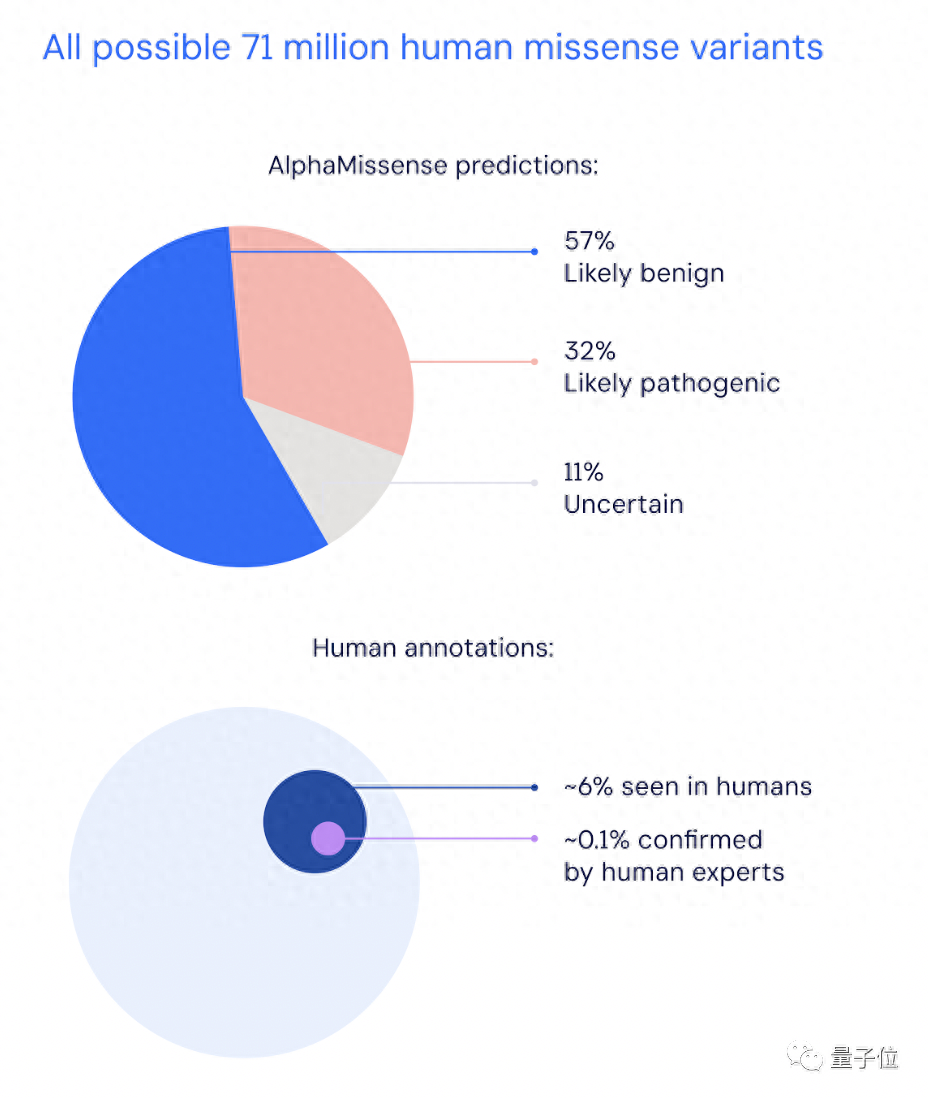
已对7100万错义突变进行分类
鱼羊 发自 凹非寺
量子位 | 公众号 QbitAI
基因突变会如何影响人类健康,很大程度上还是个未解之谜。
不过现在,人类可以借AI之力来研究这件事儿了:
谷歌DeepMind在AlphaFold的基础上,训练出了专门预测人类基因组中错义突变致病性的AlphaMissense。
论文今天登上Science。
“错义突变”是DNA序列的一种非同义替换。简单来说,就是DNA原本的字母(碱基对)被替换掉了。
这就意味着,对应生成的蛋白质里的氨基酸也会发生异化,这可能会让蛋白质失去原本的功能,导致疾病的产生。
AlphaMissense出山第一步,就是对全部7100万种可能的错义突变进行了分类。
结果是,这个AI成功将这些变异中的89%,分类为“可能致病”和“可能良性”。相比之下,人类专家目前的成绩是0.1%。
用AI预测基因突变致病性
一句话总结,AlphaMissense的主要能力,就是预测人类基因组中所有可能的错义突变是致病性的还是良性的。
如何做到——
AlphaMissense基于DeepMind的蛋白质结构预测模型AlphaFold打造。
研究人员用人类和灵长类变异频率数据库对AlphaFold进行了微调。具体而言,自然界中的常见变异可以视作对生物无害的变异,而未曾在数据库中出现的变异,则可视作“致病变异”训练数据。
这种训练策略,可以避免人工标注带来的偏见。
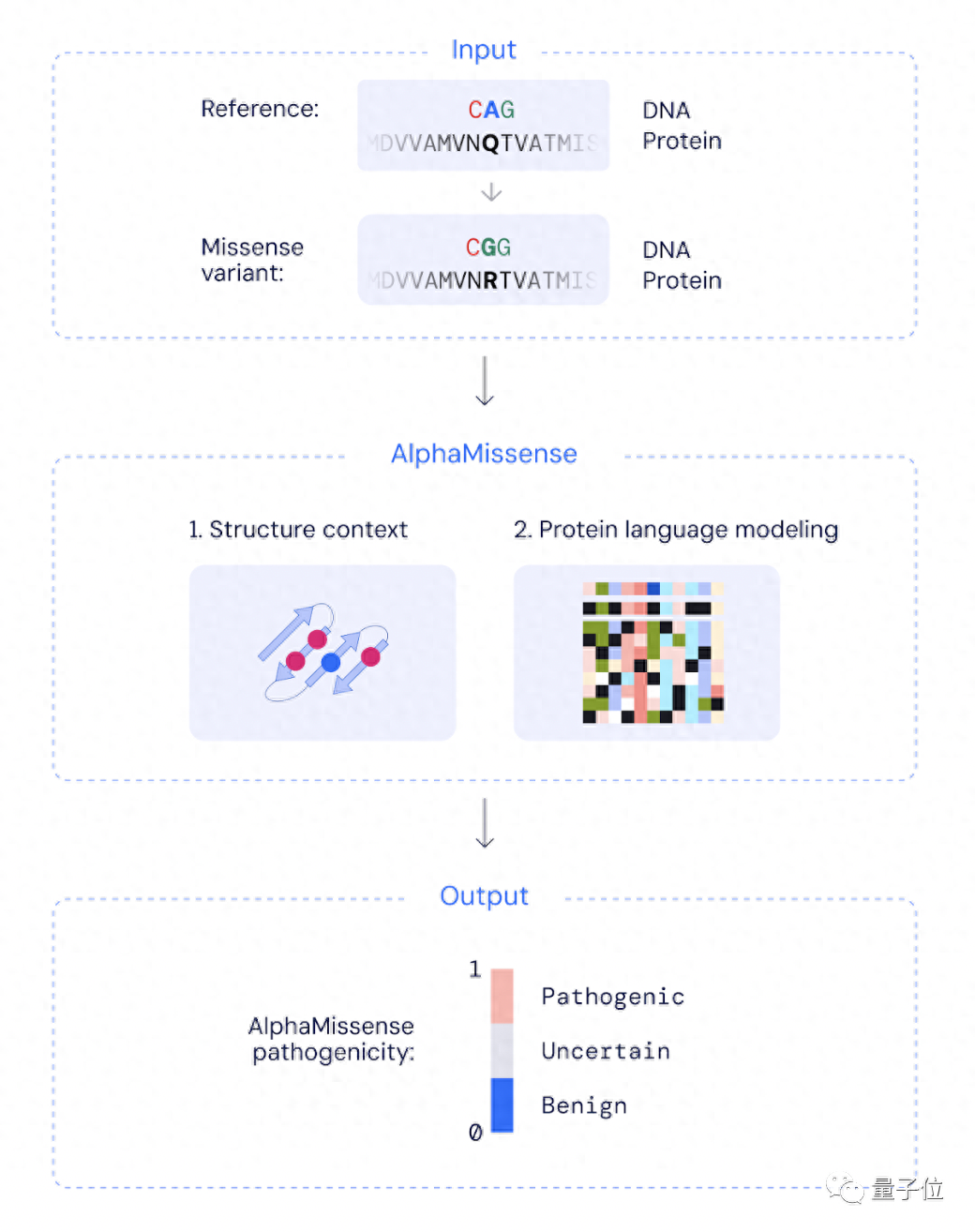
值得一提的是,AlphaMissense不能预测突变后蛋白质结构的变化和突变对蛋白质稳定性的其他影响。
在输入一种错义突变之后,AlphaMissense会结合蛋白质结构上下文和蛋白质语言模型,对突变进行一个0-1分的打分,以此大致判定这种突变会不会致病。
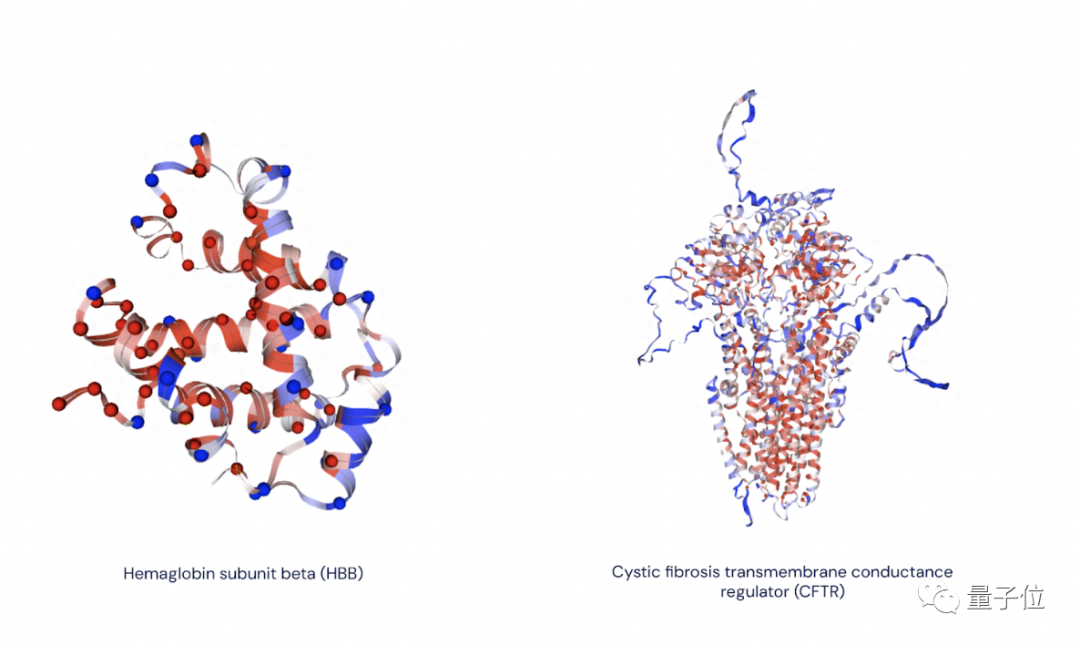
△AlphaMissense+AlphaFold效果
那么问题来了,AlphaMissense的这种分类,真的可靠吗?
研究人员对其进行了实验验证。
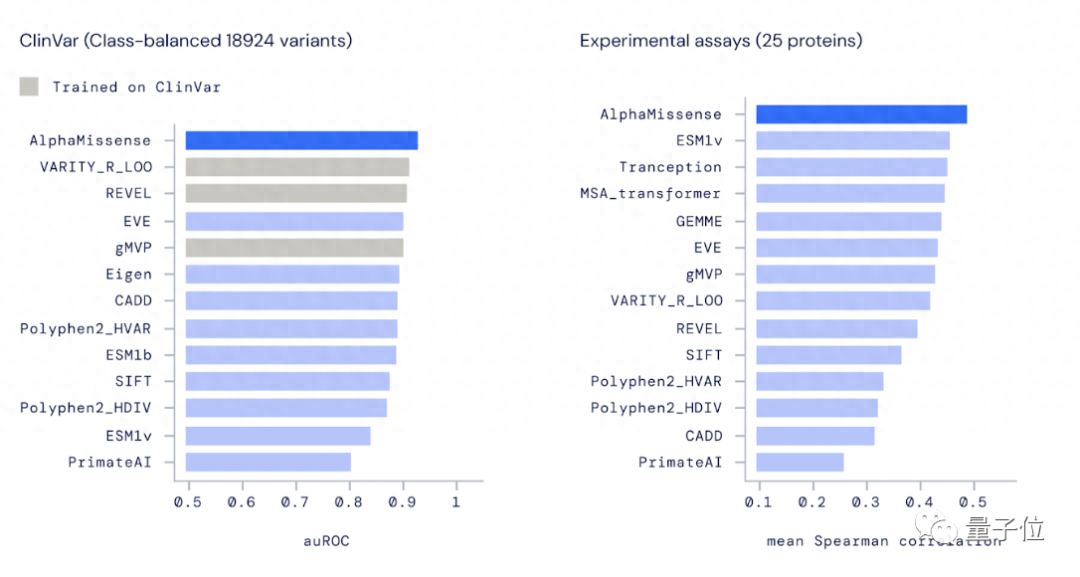
在遗传学权威数据库ClinVar上,AlphaMissense展现出了比其他计算方法更强大的分类性能。
在18924个变异数据中,AlphaMissense的ROC曲线下面积(auROC)达到0.94。这个数字越接近于1,就代表模型越能正确地区分正样本和负样本。
值得注意的是,上图中,以灰色显示的计算方法是在ClinVar上训练的,可能存在过拟合。
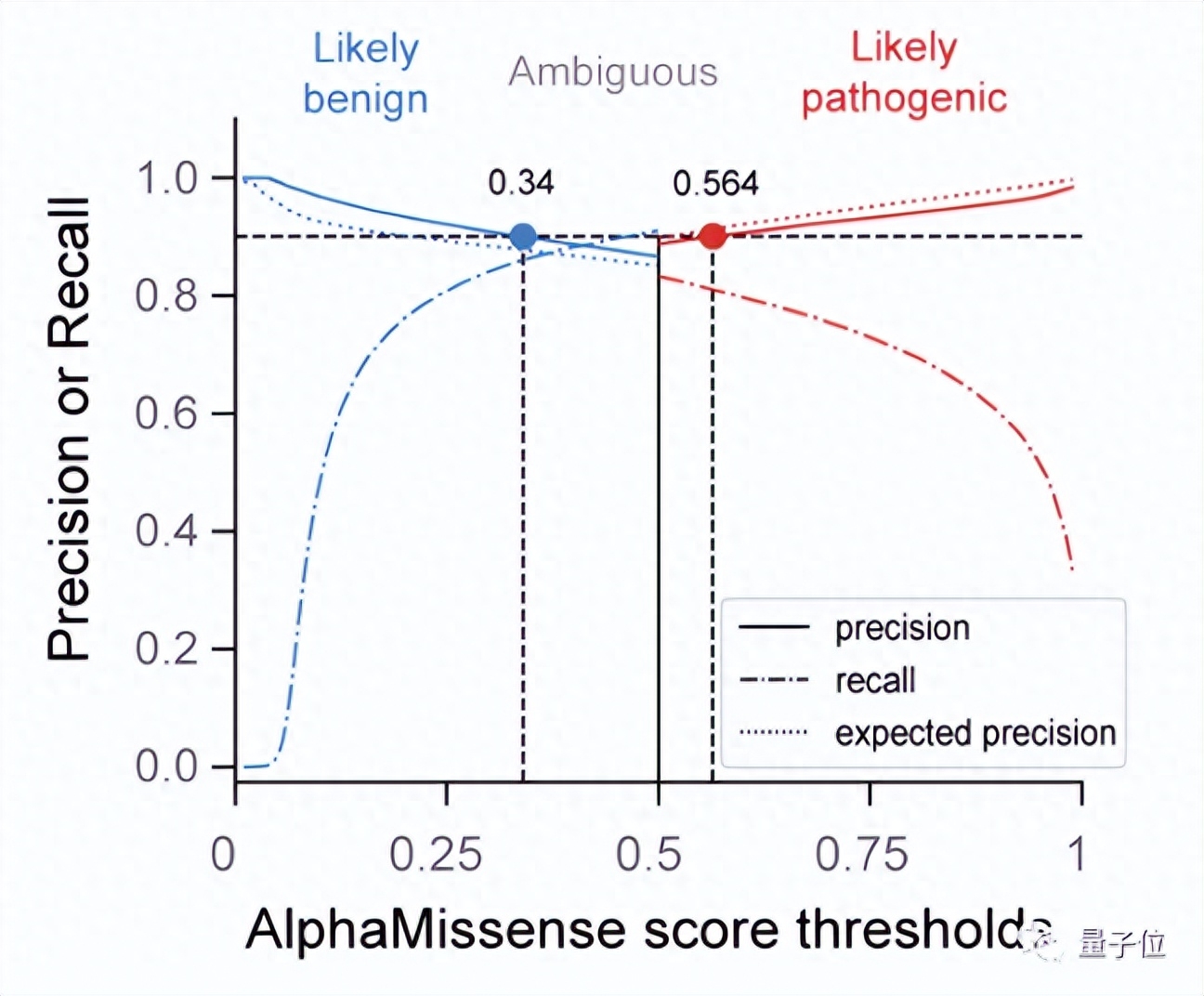
在预测准确率方面,AlphaMissense也达到了SOTA。通过调节分类阈值,AlphaMissense给出的“可能致病”和“可能良性”分类,期望精确度都达到了90%。
DeepMind表示:
我们期待看到AlphaMissense帮助解决基因组学和生物科学中悬而未决的问题。
为此,他们已经把AlphaMissense的预测结果和模型代码开源了出来。
另外,DeepMind还分享了19000多种人类蛋白质中所有可能的2.16亿个单氨基酸序列置换预测数据集。
参考链接:
[1]论文地址:https://www.science.org/doi/10.1126/science.adg7492
[2]https://www.deepmind.com/blog/alphamissense-catalogue-of-genetic-mutations-to-help-pinpoint-the-cause-of-diseases
[3]https://github.com/deepmind/alphamissense
— 完 —
- 马斯克冲刺机器人量产,果断停产特斯拉豪华车型!2026年资本支出将“非常大”2026-01-29
- 那个用半成品刷爆SOTA的Qwen3超大杯推理版,现在正式上线2026-01-26
- “DeepSeek-V3基于我们的架构打造”,欧版OpenAI CEO逆天发言被喷了2026-01-26
- 斯坦福「返老还童」新研究:无需干细胞,逆转关节损伤和老化2026-01-25